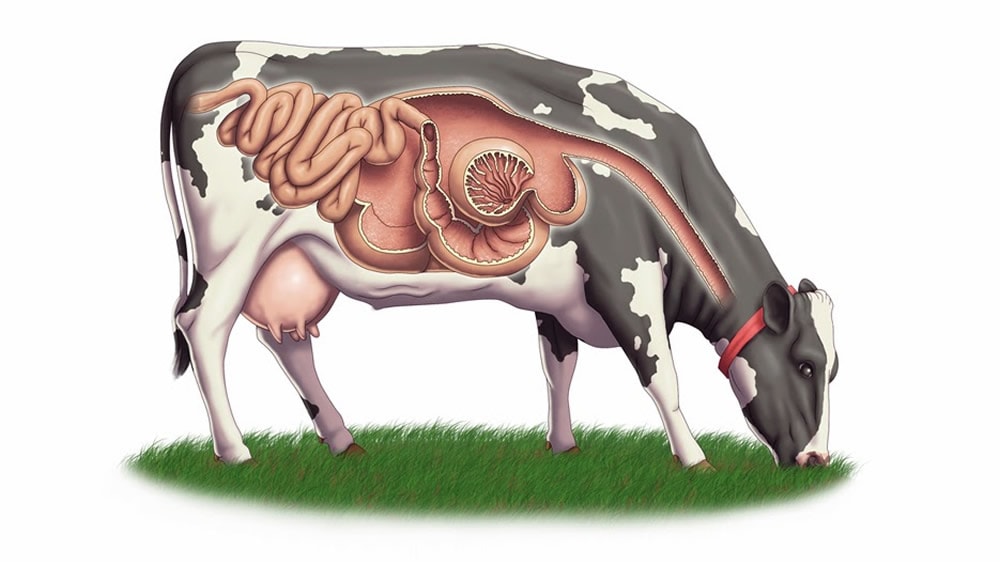
Conocidos como rumiantes, en el reino animal los bovinos, caprinos y ovinos, entre otros, tienen la capacidad de convertir pasto en proteína animal de alto valor. Para que esto sea posible, la flora microbiana presente en el rumen fermenta el alimento y facilita la digestión de la materia vegetal rica en celulosa y lignina. Como resultado de este proceso, los animales emiten metano –uno de los gases de efecto invernadero–.
Comprender las funciones de los microorganismos que integran el rumen, resulta esencial para el desarrollo de dietas más eficientes y, a su vez, ayudaría a minimizar las emisiones de gases de efecto invernadero (GEI). Por esto, el consorcio internacional Hungate1000, integrado por más de 55 científicos de 19 organismos de investigación de nueve países, analizó y catalogó los genomas de 410 bacterias cultivadas y arqueas –organismos unicelulares– presentes en el rumen de varias especies animales.
Del estudio, recientemente publicado en la revista científica Nature Biotechnology, participaron investigadores del Instituto de Biotecnología y del Instituto de Patobiología, ambos del Centro de Investigación en Ciencias Veterinarias y Agronómicas (CICVYA) del INTA.
Silvio Cravero, investigador del Instituto de Biotecnología del INTA, aseguró que la investigación “es un verdadero impulso a la biología del microbioma del rumen, ya que no solo permitió secuenciar los genomas de bacterias y arqueas que viven allí, sino determinar que 134 son las mismas que se encuentran en el intestino humano”.
Además de la caracterización de 410 microorganismos, el consorcio internacional analizó 91 genomas de interés, lo que elevó a 501 el número de organismos secuenciados –480 bacterias y 21 arqueas–. De esta manera, se estima que se analizó aproximadamente el 75 % de los géneros bacterianos presentes en el rumen.
Además de la caracterización de 410 microorganismos, analizaron 91 genomas de interés, lo que elevó a 501 el número de organismos secuenciados –480 bacterias y 21 arqueas–.
“Este trabajo contribuirá a conocer la diversidad y el funcionamiento de las bacterias y arqueas presentes en el rumen y aportará al diseño y monitoreo de estrategias de mitigación de metano en los sistemas de producción animal”, destacó Cravero y agregó: “Será una línea de base para el estudio de microbiotas de otras especies animales”.
La colección de microorganismos y sus genomas, “constituyen un recurso biológico que se puede utilizar para avanzar en el estudio de secuencias de genes con propiedades a escala industrial y en la comprensión de la fisiología ruminal”, destacó Cravero y agregó: “Esto permitirá encontrar un equilibrio entre consumo, producción y reducción de las emisiones de gases de efecto invernadero”.
María Esperanza Ceron Cucchi, del Instituto de Patobiología del INTA e integrante del consorcio internacional Hungate1000, explicó que “bacterias, protozoos, arqueas, hongos y bacteriófagos conforman la microbiota rumial cuya principal función es la de digerir enzimáticamente el alimento vegetal”.
De acuerdo con Ceron Cucchi, “la microbiota contribuye ampliamente a la promoción de la salud, productividad e inmunidad del animal. Es un campo de investigación que está en pleno auge en el mundo”.
“Por su función, estos microorganismos residentes en el tracto gastrointestinal determinan la extracción de nutrientes y participan en la regulación de su balance energético”, expresó la especialista del INTA.
De acuerdo con el artículo Cultivation and sequencing of rumen microbiome members from the Hungate1000 Collection, la fermentación entérica en rumiantes libera unos 125 millones de toneladas de metano a la atmósfera al año y es considerada la fuente antropogénica más importante de emisión de este gas de efecto invernadero.
En 2012, cuando el consorcio inició los estudios, solo 14 genomas microbianos estaban disponibles para su estudio.
Comprender las funciones de los microorganismos que integran el rumen es esencial para el desarrollo de dietas más eficientes y, a su vez, ayudaría a minimizar las emisiones de GEI.
Un catálogo para futuros desarrollo
Los aislamientos generados permitirán avanzar no solo en investigación orientada a la producción de biocombustibles, sino también de alimentos para animales, aditivos, antibióticos y aceites esenciales, así como incursionar en la generación de vacunas contra arqueas metanógenas, debido a que con la secuencia del genoma se puede identificar la proteína candidata para la vacuna. Tampoco descartan las aplicaciones en humanos.
El Laboratorio de Microbiología del Rumen del Instituto de Patobiología del INTA –único representante de Sudamérica en el grupo internacional– generó los primeros datos relacionados a la caracterización microbiana del rumen en camélidos sudamericanos.
Recientemente, el Instituto de Biotecnología inició colaboraciones con laboratorios de Nueva Zelanda para expresar en levaduras proteínas de arqueas metanogénicas. Serán evaluadas como inmunógenos en el diseño de vacunas destinadas a mitigar la emisión de gas metano derivado de la producción animal.
Los animales rumiantes -bovinos, caprinos y ovinos, entre otros- son capaces de convertir materia vegetal lignocelulósica en proteína animal de alto valor como carne, leche y fibra.
El rumen está formado por una comunidad de microorganismos o microbiota compleja constituida por bacterias, protozoos, fagos, arqueas y hongos. Se trata de un microbioma con alta densidad y diversidad microbiana con unos 10 mil millones de microorganismos por mililitros.
Como consecuencia del proceso de fermentación, se generan compuestos como ácidos grasos de cadena corta (acético, propiónico y butírico son los que se producen en mayor cantidad en la fermentación de los alimentos en el rumen) que son absorbidos y utilizados como fuente de energía por el animal.
Durante el proceso, también se generan moléculas simples como amonio, dióxido de carbono, hidrógeno y metano, este último reconocido entre los principales gases de efecto invernadero que contribuyen al cambio climático.
Si bien el ecosistema ruminal se estudia desde hace varios años, el conocimiento sobre la microbiota involucrada en la degradación de material lignocelulósico y de la formación del gas metano es limitado.
Es necesario comprender que un animal, un humano, una planta e incluso un insecto para desarrollarse requieren estar asociados a comunidades bacterianas determinadas, lo que lleva a definir el término holobionte. Donde cada uno conforma una unidad ecológica en conjunto con sus microbiomas y evolucionan juntos.
Del estudio, participaron investigadores del Instituto de Biotecnología y del Instituto de Patobiología y Biotecnología, ambos del Centro de Investigación en Ciencias Veterinarias y Agronómicas (CICVYA).
De la publicación en la revista Nature participaron científicos de Nueva Zelanda, Estados Unidos, Canadá, Australia, Japón, Reino Unido, Francia, Arabia Saudita y Dinamarca junto con integrares del proyecto Hungate1000 (cuyo nombre se debe al objetivo de llegar a estudiar 1.000 aislamientos microbianos ruminales).
El consorcio, liderado por los investigadores del Centro de Investigación en Pasturas del AgResearch, de Nueva Zelanda, recibió financiamiento del gobierno de ese país y del Departamento de Energía de los Estados Unidos, mediante el Joint Genome Institute (JGI). Además, tuvo el auspicio de la Alianza Global para la Investigación de Gases de Efecto Invernadero en la Agricultura (conocida como Global Research Alliance).