1. Introducción
La salmonelosis transmitida por alimentos es una de las principales preocupaciones de salud pública a escala global1. En este ámbito, México no es la excepción, pues la Salmonella no tifoidea (asociada con los animales de abasto), ocupó el segundo lugar como agente de infecciones humanas en 2016, según datos de la Secretaría de Salud (http://www.epidemiologia.salud.gob.mx/anuario/html/anuarios.html).
Dado que en el país no se dispone de un sistema activo de vigilancia epidemiológica, no es posible conocer los alimentos involucrados en las infecciones de origen alimentario. No obstante, investigaciones recientes de México y de otros países demuestran que los bovinos constituyen un importante reservorio de Salmonella spp2-5.
En casi la totalidad de los estudios antes referidos, se sugiere que la presencia de Salmonella, en animales aparentemente sanos y en los productos que de ellos se derivan, constituye un riesgo para la salud. Además, se considera que el riesgo es aun mayor cuando las cepas aisladas son capaces de resistir a varios antibióticos empleados para tratar infecciones humanas. Sin embargo, son muy escasos los estudios que profundizan en los determinantes genéticos asociados con la habilidad para causar infecciones y los mecanismos de resistencia a antimicrobianos de las cepas obtenidas. Lo anterior es vital para identificar los factores que favorecen la persistencia de Salmonella en la cadena alimenticia y, por ende, para estimar los riesgos que estas cepas representan para la inocuidad de los alimentos y la salud pública.
Para atender esta problemática, el grupo de investigación del Laboratorio de Ciencia de la Carne (Facultad de Medicina Veterinaria y Zootecnia, Universidad Nacional Autónoma de México), abrió recientemente una línea de investigación sobre estudios genómicos de las poblaciones de Salmonella presentes en la cadena de bovinos de carne en México. Con ello se espera contribuir a mejorar el conocimiento de las bases genéticas asociadas con el establecimiento, la persistencia y la diseminación de Salmonella en la cadena alimenticia y, por ende, a identificar medidas de control para minimizar el riesgo asociado con este patógeno, tanto en México como a escala global. En el presente trabajo se resumen los resultados más relevantes de los proyectos realizados recientemente por nuestro equipo de investigación.
2. Resumen de la metodología de investigación empleada
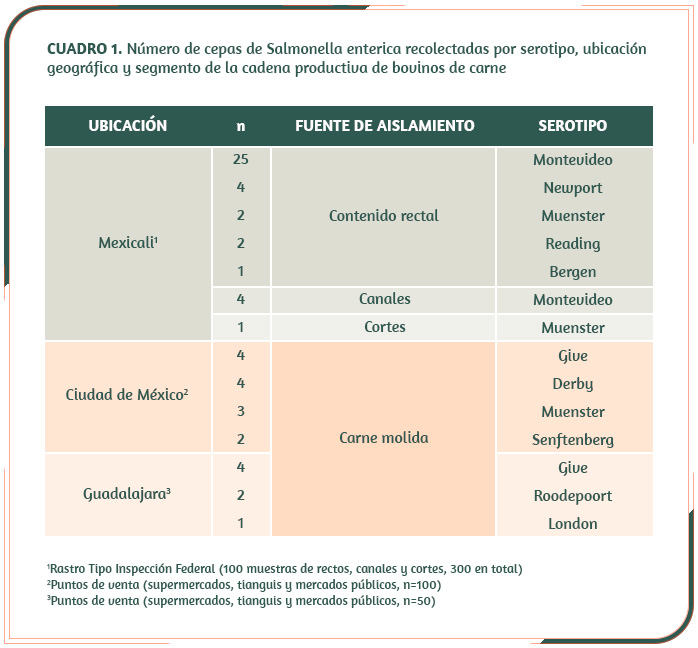
El Cuadro 1 describe el panel de 59 cepas, obtenido en investigaciones realizadas en 2013. Se incluyeron cepas de múltiples serotipos y ubicaciones geográficas distantes, todas asociadas con la especie bovina y obtenidas en diferentes segmentos de la cadena productiva: 39 en un rastro Tipo Inspección Federal ubicado en el norte de México y las 20 restantes en puntos de venta de las ciudades de México y Guadalajara.
La detección y el aislamiento de Salmonella se realizó conforme a la Norma Oficial Mexicana vigente en 20136. Las muestras presuntivas fueron confirmadas mediante la técnica de reacción en cadena de la polimerasa (PCR), utilizando el gen invA (284 pb), según métodos descritos previamente7. Además, se determinó el perfil de resistencia a antimicrobianos, por el método de Kirby-Bauer8, utilizando un panel de 14 antibióticos incluidos en la lista de medicamentos esenciales de la Organización Mundial de la Salud (OMS)9.
El genoma completo de las cepas se secuenció en un equipo Illumina MiSeq, utilizando el kit Nextera XT version 2 para preparar la biblioteca de ADN, con un inserto de 250 pb y una profundidad estimada mínima de 30X. Los datos de secuenciación se analizaron y depuraron con los programas FastQC y Trimmomatic, respectivamente. Por último, los genomas fueron ensamblados de novo con el algoritmo Spades10 y anotados en el servidor RAST11.
3. Los bovinos como reservorio de Salmonella enterica
Estos estudios mostraron que Salmonella puede encontrarse en altas frecuencias en las heces de bovinos aparentemente sanos. Lo anterior podría deberse al establecimiento del patógeno en granjas; o bien, al incremento en la excreción fecal del mismo, que suele ocurrir debido al estrés fisiológico de los animales durante el transporte y el manejo ante mortem12. Cabe destacar, no obstante, que las intervenciones antimicrobianas que se aplican en el rastro (retiro de materia fecal visible y tratamiento con vapor) reducen considerablemente la contaminación en canales y cortes, en los cuales el patógeno se presentó en niveles similares a los que se observan en países desarrollados13.
4. La producción animal parece favorecer poblaciones de Salmonella con perfiles genómicos adaptados para sobrevivir en ciclos continuos de infección fecal-oral
La tipificación por multi-locus basada en la secuenación completa del genoma (wgMSLT, por sus siglas inglesas), permite determinar, con un alto poder discriminatorio, la diversidad genética de las poblaciones. En el caso de Salmonella, el wgMSLT agrupa las cepas considerando la similitud en la secuencia de 21,065 clusters de genes obtenidos a partir del pangenoma de 537 cepas. Este análisis, realizado en el servidor de Enterobase (http://enterobase.warwick.ac.uk), demuestra el origen fecal de la Salmonella presente en carne de bovino, debido a que aislamientos de canales y de cortes son prácticamente idénticos a los presentes en el contenido rectal (ver agrupación de cepas Montevideo y Muenster, Figura 1 y Cuadro 1). Además, evidenció la diseminación clonal de cepas en el país. Por ejemplo, aislamientos Give y Muenster, obtenidos en zonas separadas entre sí por 500-2000 km, resultaron prácticamente idénticos.
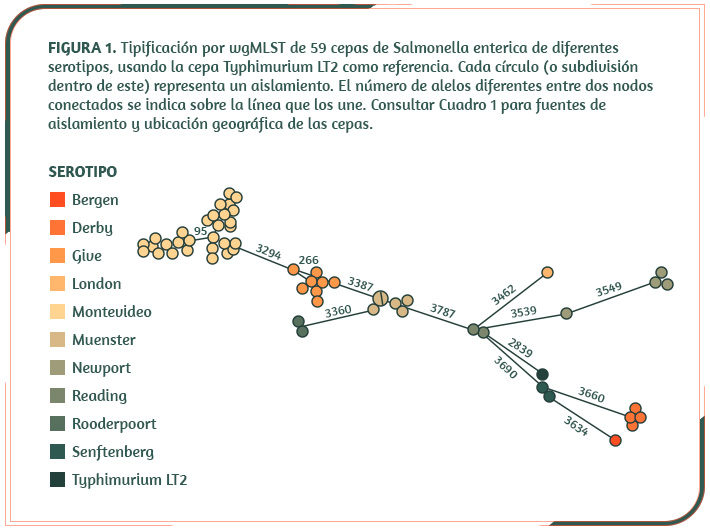
Es interesante notar que las cepas que conforman el subgrupo mayoritario (lado izquierdo de la Figura 1), representan el 76% de los aislamientos, cercanos entre sí a pesar de ser de diferentes serotipos y provenir de fuentes no relacionadas epidemiológicamente. Además, éstas muestran una evolución divergente con respecto al subgrupo minoritario, cuyos representantes se asemejan a Typhimirum LT2.
Dado que todas las cepas son de origen bovino, este patrón de diversidad genética sugiere que la presión evolutiva del nicho ecológico estudiado pudo haber favorecido la emergencia de genotipos menos virulentos hacia el bovino, creando “genovariedades” especializadas para sobrevivir en el mismo. Estas “genovariedades” adaptadas pueden persistir y diseminarse de forma más eficiente en la cadena productiva, por lo que resultan más preocupantes para la inocuidad de los alimentos y la salud pública.
5. La adaptación a los bovinos no excluye el riesgo de infecciones humanas
Aunque las cepas de Salmonella estudiadas son capaces de infectar los bovinos sin provocar signos visibles de enfermedad, éstas poseen un repertorio conservado de genes de virulencia, por lo que su potencial para provocar infecciones humanas no debe descartarse.
Por ejemplo, aunque ninguna de las cepas presentó genes asociados con fenotipos de alta virulencia (ej. spvRABCD, pefABCD, rcK, mig-5), se observó que los factores asociados con adherencia intestinal (ej. fimACDFHIWYZ, csgABCEFG), captación de hierro (fur, iroBCDE, fepBCDEG, fhuBCD, exbBD, tonB) y activación y/o regulación de los mecanimos de invasión y de supervivencia intracelular (phoPQ, fur, mgtBC, rpoS, mig-14) están prácticamente inalterados (Figura 2).
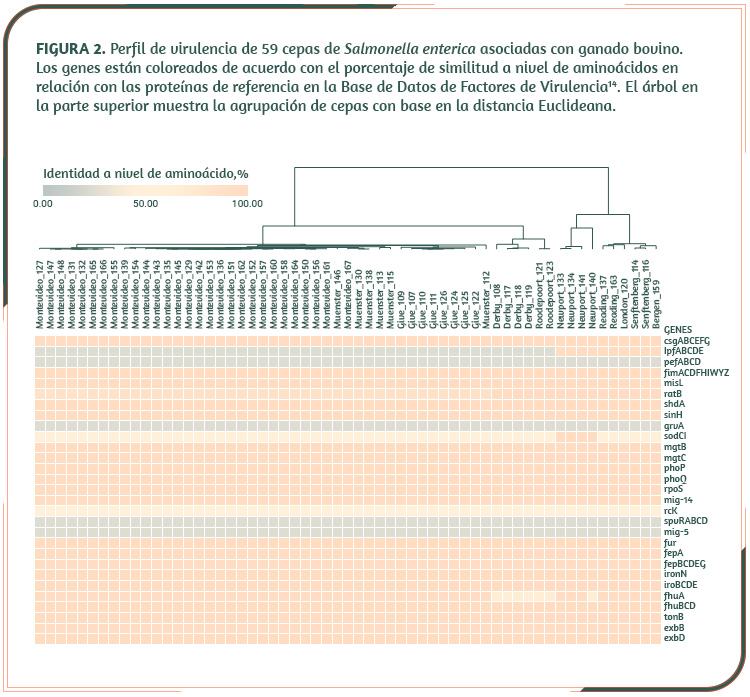
También se estudiaron las islas de patogenicidad (SPI, por sus siglas inglesas), las cuales se sabe que juegan un papel importante en la patogenia de Salmonella. Salvo algunas diferencias en genes de función desconocida, las SPI 1 a 5 están altamente conservadas en las cepas bajo estudio.
6. Preocupante perfil de resistencia a antimicrobianos
Las pruebas de susceptibilidad realizadas en un subgrupo de 44 cepas, demostraron que 66% de estas (casi 7 de cada 10) resisten dos o más clases de antibióticos (Figura 3), por lo que se consideran multi-resistentes (MDR). El perfil de MDR incluye varios de los fármacos considerados como medicamentos esenciales por la OMS9.
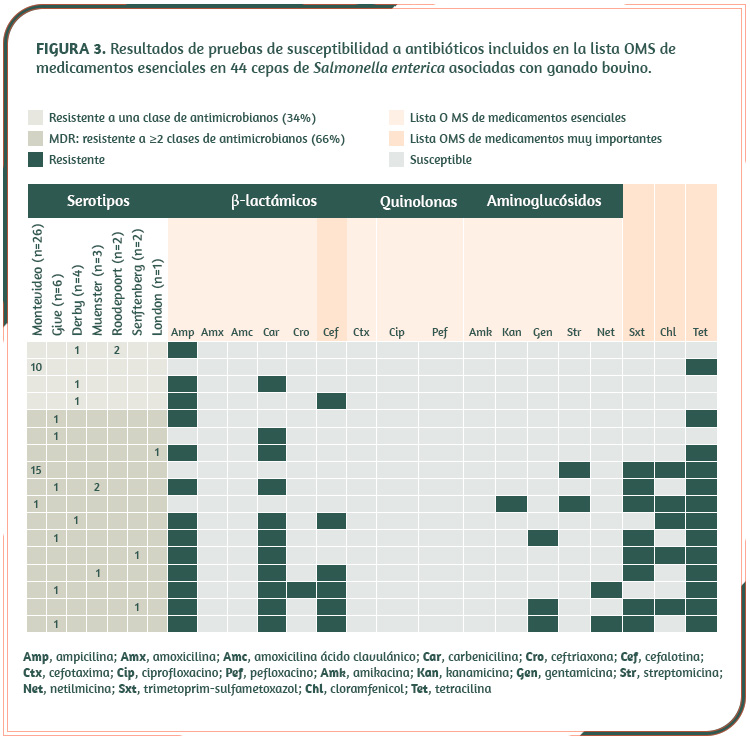
En general, el genotipo de resistencia explicó los fenotipos observados, excepto en betalactámicos, tetraciclina y quinolonas (Figura 4). En los dos primeros, se observó resistencia en algunas cepas, a pesar de que éstas no tenían genes específicos que explicaran tal fenotipo. Sin embargo, esto puede deberse a la presencia de bombas de eflujo, las cuales se sabe que pueden compensar la falta de genes de resistencia15.
En el caso de las quinolonas, resulta preocupante la detección de genes de resistencia asociados con plásmidos (PMQR, por sus siglas inglesas). Aunque ningún aislamiento mostró resistencia a quinolonas, se sabe que los PMQR confieren bajos niveles de resistencia, inferiores al punto de quiebre utilizado como referencia en las pruebas de susceptibilidad. Esto podría resultar en niveles de resistencia superiores si el patógeno es desafiado con quinolonas en concentraciones terapéuticas16.
También se detectaron genes de resistencia a fosfomicina y azitromicina, ambos en la lista OMS de medicamentos esenciales. Aunque éstos no fueron incluidos en las pruebas de susceptibilidad, varias cepas codifican factores de resistencia para ambos, por lo que existe el potencial de que el perfil de resistencia sea mucho más amplio que el observado.
Por último, se observó una amplia diseminación del gen qacEDelta1, que confiere resistencia a los compuestos de amonio cuaternario17. Esto evidencia la necesidad de revisar las prácticas de aplicación y sobre todo de rotación de desinfectantes en la producción de alimentos, con el fin de evitar la selección de cepas resistentes y, de esta forma, contribuir a su persistencia y diseminación en el ambiente.
En resumen, los resultados de resistencia a antimicrobianos evidencian la importancia de estos genes para la supervivencia y la diseminación del patógeno. Sin duda, su presencia en el genoma obedece al continuo estrés selectivo a que es sometida la bacteria, producto del empleo de agentes antimicrobianos en producción animal. La presencia en puntos de venta de “supercepas” capaces de resistir entre 4 y 7 antibióticos diferentes así lo confirma.

7. Conclusiones
La secuenciación completa del genoma de cepas de Salmonella de origen no clínico, obtenidas a lo largo de la cadena de bovinos de carne en México, evidencia que éstas poseen una maquinaria de virulencia prácticamente intacta, así como un vasto repertorio de genes de resistencia a antibióticos. Lo anterior reafirma la necesidad de mejorar el control del patógeno desde las granjas, que es el segmento desde el que ocurre la diseminación. Por ejemplo, la aplicación de programas de monitero de infecciones subclínicas podría favorecer la detección oportuna y, por ende, el aislamiento de diseminadores potenciales.También se debe señalar la necesidad de optimizar el uso de antibióticos y desinfectantes en la producción de alimentos, para evitar que las prácticas zootécnicas favorezcan la emergencia de “supercepas”, mismas que pueden representar un serio desafío para la salud pública.
8. REFERENCIAS
- WHO. 2015. WHO estimates of the global burden of foodborne diseases. Foodborne disease burden epidemiology reference group 2007-2015. World Health Organization, <http://www.who.int/foodsafety/areas_work/foodborne-diseases/ferg/en/>. Consultado: December 11, 2016.
- Eguale, T. et al. Fecal prevalence, serotype distribution and antimicrobial resistance of Salmonellae in dairy cattle in central Ethiopia. BMC microbiology 16, 20, (2016).
- Cabrera-Diaz, E. et al. Occurrence, serotype diversity, and antimicrobial resistance of salmonella in ground beef at retail stores in Jalisco state, Mexico. J Food Prot 76, 2004-2010, (2013).
- Varela-Guerrero, J. A., Talavera-Rojas, M., Gutierrez-Castillo Adel, C., Reyes-Rodriguez, N. E. & Vazquez-Guadarrama, J. Phenotypic-genotypic resistance in Salmonella spp. isolated from cattle carcasses from the north central zone of the State of Mexico. Trop Anim Health Prod 45, 995-1000, (2013).
- Yin, M. et al. Prevalence and characterization of Salmonella enterica serovar in retail meats in market place in Uighur, Xinjiang, China. Food Control 64, 165-172, (2016).
- SSA. (Diario Oficial de la Federación. 15 de agosto de 1994, 1994).
- Rahn, K., De Grandis, S. A., Clarke, R. C., Curtiss, R. & Gyles, C. L. Amplification of an invA gene sequence of Salmonella typhimurium by polymerase chain reaction as a specific method of detection of Salmonella. Molecular and cellular probes 6, 271-279, (1992).
- Bauer, A. W., Kirby, W. M., Sherris, J. C. & Turck, M. Antibiotic susceptibility testing by a standardized single disk method. Am J Clin Pathol 45, 493-496, (1966).
- WHO. WHO list of critically important antimicrobials for human medicine 5th revision. (2017). <http://who.int/foodsafety/publications/antimicrobials-fifth/en/>.
- Bankevich, A. et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing. J Comput Biol 19, 455-477, (2012).
- Overbeek, R. et al. The SEED and the Rapid Annotation of microbial genomes using Subsystems Technology (RAST). Nucleic Acids Research 42, D206-D214, (2014).
- Gopinath, S., Carden, S. & Monack, D. Shedding light on Salmonella carriers. Trends in microbiology 20, 320-327, (2012).
- Rhoades, J. R., Duffy, G. & Koutsoumanis, K. Prevalence and concentration of verocytotoxigenic Escherichia coli, Salmonella enterica and Listeria monocytogenes in the beef production chain: a review. Food Microbiol 26, 357-376, (2009).
- Chen, L., Zheng, D., Liu, B., Yang, J. & Jin, Q. VFDB 2016: hierarchical and refined dataset for big data analysis–10 years on. Nucleic Acids Res 44, D694-697, (2016).
- Hur, J., Jawale, C. & Lee, J. H. Antimicrobial resistance of Salmonella isolated from food animals: A review. Food Research International 45, 819-830, (2012).
- Strahilevitz, J., Jacoby, G. A., Hooper, D. C. & Robicsek, A. Plasmid-mediated quinolone resistance: a multifaceted threat. Clin Microbiol Rev 22, 664-689, (2009).
- Wassenaar, T. M., Ussery, D., Nielsen, L. N. & Ingmer, H. Review and phylogenetic analysis of qac genes that reduce susceptibility to quaternary ammonium compounds in Staphylococcus species. Eur J Microbiol Immunol (Bp) 5, 44-61, (2015).